PCR (Polymerase Chain Reaction)
La PCR ou réréaction de polymépolymérisation en chaine, est un des élééléments favoris de la boiteboîte àà outils des biologistes au laboratoire.
A la fois trètrès simple et d'une grande éléélégance, elle permet d'amplifier des séséquences d'acides nuclénucléiques variévariés.
Avant de commencer
Comme toutes les sciences, la biologie àà sa propre langue, donc pour se comprendre il nous faut dédéfricher un peu le vocabulaire.
Voici quelques clésclefs :
-les acides nuclénucléiques sont les supports de l'information génégénétique. Ils sont constituéconstitués de 4 briques universelles diffédifférentes nomménommées nuclénucléotides (A, C, G et T/U). AgencéAgencées en séséquence àà brin unique (ARN) ou àà double brin (ADN), ces briques repréreprésentent une information qui peut êêtre recopiérecopiée (c'est le principe de la PCR) ou bien décodédécodée par une machinerie spéspécifique àà base d'enzymes. Ces séséquences parfois extrêextrêmement longues sont fragiles et souvent enrouléenroulées de manièmanière poussépoussée sur elles-mêmêmes et autour de bobines protéprotéiques.
-la dédénaturation est un processus qui vise àà linélinéariser au mieux les séséquences d'acides nuclénucléiques pour les rendre accessibles aux enzymes. Dans le cas des ADN double brins, ces derniers s'éécartent partiellement en deux brins uniques reliéreliés par quelques points (comme les anneaux d'une chaines), donnant accèaccès àà chaque brin individuellement.
-l’l’appariement est le principe de complémentaritécomplémentarité des 4 diffédifférents nuclénucléotides dans les séséquences double brin d'ADN. Il se fait d'un brin àà l'autre entre nuclénucléotides complécomplémentaires qui se font face. C'est assez simple car il n'y a en fait qu'un appariement possible pour chaque nuclénucléotide : le A avec le T, et le C avec le G ! Donc par exemple pour une courte séséquence monobrin GATTACA, le brin complécomplémentaire est CTAATGT.
-les enzymes sont des machines àà base de protéprotéines hyper-spécialiséspécialisées, extrêextrêmement efficientes et trètrès diversifiédiversifiées. Elles sont génégénéralement spéspécifiques àà un petit nombre de molémolécules (nomménommé substrat) sur lesquelles elles se fixent pour réréaliser des opéopérations simples mais préprécises, avec un faible taux d'erreur. Par exemples elles peuvent tronçtronçonner une molémolécule, en fusionner deux bout àà bout, gégérer du stockage, libélibérer de l'éénergie, ou dans le cas de la TAQpolyméTAQpolymérase qui nous intéintéresse ici, réréaliser la copie d'une séséquence.
-la La TAQpolyméTAQpolymérase est l'enzyme utiliséutilisée pour réréaliser la PCR. Elle se lie spéspécifiquement aux séséquences d'acides nuclénucléiques monobrin et cherche a recrérecréer le brin manquant.
Les éétapes
En quoi consiste la PCR : une enzyme nomménommée polymépolymérase va venir faire des trètrès nombreuses copies d'une séséquence modèmodèle d'acides nuclénucléiques.
On réréalise plusieurs éétapes àà des tempétempératures diffédifférentes dans un appareil appeléappelé thermocycleur. Le mémélange réréactionnel (Taq polymépolymérase, Mg2+ et parfois MgCl2, amorces, les 4 dNTP en excèexcès (A, T, C, G) et l’l’extrait d’d’ADN cible, tampon réréactionnel ) est placéplacé dans des microtubes et est soumis, plusieurs dizaines de fois, àà des cycles successifs. Le thermocycleur permet la programmation de chaque éétape des cycles. La réréaction PCR dure quelques heures (2 àà 3 heures pour une PCR de 30 cycles).
Le nombre de cycles est typiquement inclus entre 25-35 cycles d'amplification. Plus on effectue de cycles, plus on augmente le risque d'avoir des contaminants au sein de la réréaction. Il est donc conseilléconseillé d'effectuer le nombre minimum de cycles nénécessaire afin de synthésynthétiser la quantitéquantité de produit désirédésirée.
Le protocole diffèdiffère en fonction du type de PCR àà effectuer :
PCR classique :
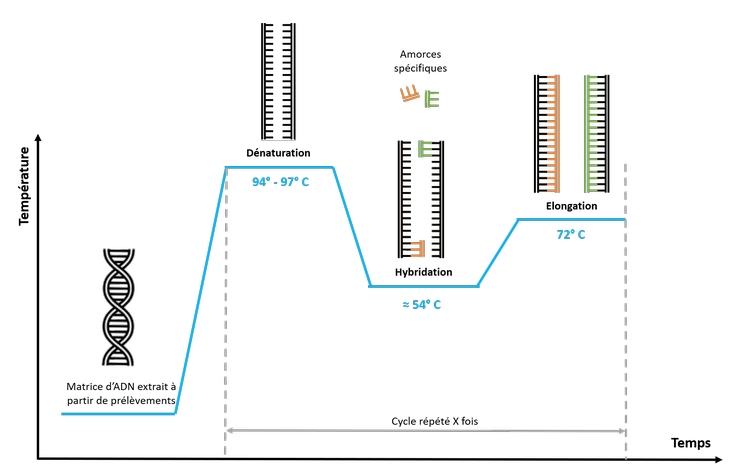
1 : ÉÉtapes du cycle PCR
DéDénaturation thermique de l'ADN àà 95°95°C : permet de rompre les liaisons hydrogèhydrogène afin d'obtenir des matrices simple brin.
Hybridation des amorces àà 50°50°C-65°65°C : Les deux amorces contenues en large excèexcès s'hybrident lorsqu'elles rencontrent les séséquences complécomplémentaires. La tempétempérature permet aux liaisons de se reformer. Ainsi, plus la tempétempérature d'hybridation est levélevée, plus l'hybridation est sésélective et spéspécifique.
ÉÉlongation àà 72°72°C. La Taq polymépolymérase catalyse la réréplication àà partir des ADN monocatémonocaténaires amorcéamorcés de façfaçon sésélective (sélectivitésélectivité qui dédécoule du choix des amorces).
Exemple de conditions de cycles pour une Taq polymépolymérase.
| Temps (min) |
Cycles |
||
| 2 |
1 |
||
| 0,5 |
25 |
||
| Hybridation |
0,5 |
- |
|
| Amplification |
1 |
- |
|
| Amplification finale |
5 |
1 |
|
| Trempage |
1 |
2 : ÉlectrophorèÉlectrophorèse sur gel d'agarose : Ce principe repose sur l'attraction des acides nuclénucléiques chargéchargés nénégativement sous l'effet d'un champ éélectrique. Les acides nuclénucléiques migrent plus ou moins loin àà travers le gel en fonction de la masse des molémolécules (plus elles migrent loin plus elles sont petites car le maillage du gel se rétrérétrécit plus on se rapproche du pôpôle +)
3 : RévéRévélation : L'ADN est révélérévélé par une coloration au Bromure d'ééthidium ou un intercalant visible sous lampe UV (l'intercalant se lie aux acides nuclénucléiques et éémet une fluorescence).
PCR en temps réréel :
ÀÀ l'inverse du PCR classique, le PCR en temps réréel permet une quantification plus préprécise grâgrâce àà une mesure de l'amplification tout a long de la réréaction. On effectue la dédétection et la quantification de l'amplification àà chaque fin de cycle grâgrâce àà un signal fluorescent, un intercalant des acides nuclénucléiques (donc la valeur de la fluorescence est corrélécorrélée àà la quantitéquantité de produit amplifiéamplifié). Cette dédétection éélimine le besoin d'électrophorèélectrophorèse sur gel, ce qui permet d'ééconomiser du temps et de réréduire le potentiel de contamination.
Vu que la formation de produit peut êêtre détectédétectée dans la phase exponentielle du PCR lorsque la réréaction est la plus efficace, la quantification du matématériel de dédépart est plus sensible et préprécise.
Comparaison PCR Classique et PCR temps réréel
| PCR classique |
PCR en temps |
| Visualisation du produit |
Visualisation du produit |
| Ne mesure pas |
Permet de mesurer le nombre de copies d'ADN ou d'ARN de |
| Moins |
Plus |
| Principe basique de biologie |
Demande des |
Facteurs pouvant affecter le succèsuccès de la technique de PCR :
- QuantitéQuantité d'enzyme utiliséutilisée
- Erreur de pipettage.
- Concentration de magnémagnésium (Mg2+)
Purification d'ADN :
Actuellement, l'analyse par multiplex et PCR en temps réréel marque l'importance de la qualitéqualité de la purification de l'ADN.
ÉÉtapes de base de la purification ADN : Lyser, lier, rincer, ééluer.
| Lyse : perturbation des cellules ou tissus |
- Traitement enzymatique - Perturbation - Traitement |
| Lyse : |
- - |
| Lyse : inactiver les |
- Agents perturbateurs (ex : EDTA) - |
| Lier et Rincer : |
- - |
* Liaison et rinçrinçage : techniques de séséparation de l'ADN d'autres matématériaux cellulaires.
Extraction organique : Lorsque du phéphénol ou une mixture de phéphénol/chloroforme est mélangémélangée àà un lysat cellulaire, deux phases se forment : une phase aqueuse et une phase organique. Les parts polaires de l'ADN et divisent dans la phase aqueuse, et les protéprotéines dénaturédénaturées et d'autres dédébris cellulaires se divisent dans la phase organique
Extraction grâgrâce au sel : Les sels dédéshydratent les protéprotéines, ce qui les rendent moins hydrophiles et donc dénaturédénaturées. Les protéprotéines dénaturédénaturées perdent de leur solubilitésolubilité et préprécipitent, ces protéprotéines dénaturédénaturées et les dédébris cellulaires sont extraits par centrifugation. Les sels sont typiquement utiliséutilisés dans des protocoles incluant du chlorure de sodium, potassium, acéacétate ou acéacétate d'ammonium.
Par liaison àà un support solide : La plupart des techniques de purification d'ADN sont basébasées sur la purification des lysats bruts par une liaison sésélective àà un support solide. Ces supports incluent matrice de silice, colonnes d'ééchanges anioniques, particules magnémagnétiques. GénéGénéralement ces méméthodes sont plus rapides et plus pratiques par rapport àà d'autres techniques car elles ne requièrequièrent pas de solvants organiques et peuvent êêtre miniaturiséminiaturisées et automatiséautomatisées.
Sources : Molecular biolgy, Lab Guide : Promega
Fiche Technique de la PCR
Définition
La PCR (Polymerase Chain Reaction) est une méthode permettant de copier et d'amplifier une séquence spécifique d'ADN de manière exponentielle.
Principe
La PCR repose sur l'utilisation de cycles répétitifs de dénaturation, d'hybridation et d'extension pour amplifier une séquence d'ADN cible.
Composants
-
ADN matrice: Échantillon d'ADN contenant la séquence cible à amplifier.
-
Amorces: Courtes séquences d'ADN complémentaires aux extrémités de la séquence cible.
-
dNTPs: Nucléotides libres (dATP, dTTP, dCTP, dGTP) nécessaires pour l'élongation.
-
ADN polymérase thermostable: Enzyme qui synthétise le nouvel ADN, comme la Taq polymérase.
-
Tampon: Maintient les conditions de pH et d'ions pour l'activité optimale de l'enzyme.
-
Ions magnésium (Mg²⁺): Co-facteur essentiel pour l'activité de l'ADN polymérase.
Étapes de la PCR
-
Dénaturation (94-98°C): Séparation des brins d'ADN double hélice en simples brins.
-
Hybridation (50-65°C): Liaison des amorces aux séquences complémentaires sur les brins d'ADN simple brin.
-
Élongation (72°C): Synthèse de nouveaux brins d'ADN par l'ADN polymérase en utilisant les dNTPs.
Cycle Typique de la PCR
Un cycle de PCR comprend les trois étapes ci-dessus et est répété généralement 25-35 fois.
Applications
-
Diagnostic médical: Détection de pathogènes, mutations génétiques.
-
Recherche en génétique: Clonage de gènes, étude de mutations.
-
Médecine légale: Identification d'individus à partir de traces biologiques.
-
Études évolutives: Comparaison de séquences génétiques entre espèces.
Schéma de la PCR
Tableau Résumé des Étapes de la PCR
|
Étape |
Température (°C) |
Durée |
Description |
|---|---|---|---|
|
Dénaturation |
94-98 |
30 secondes |
Séparation des brins d'ADN double brin |
|
Hybridation |
50-65 |
30 secondes |
Liaison des amorces aux séquences cibles |
|
Élongation |
72 |
1 minute/kb |
Synthèse de l'ADN par l'ADN polymérase |
Optimisation de la PCR
-
Température des amorces: Ajustement pour une hybridation spécifique.
-
Concentration des composants: Optimisation des dNTPs, amorces, et Mg²⁺.
-
Nombre de cycles: Adaptation en fonction de la quantité d'ADN initiale et de l'application.
Variantes de la PCR
-
qPCR (PCR en temps réel): Quantification de l'ADN en temps réel.
-
RT-PCR: Amplification de l'ADN complémentaire (cDNA) à partir de l'ARN.
-
PCR multiplex: Amplification simultanée de plusieurs cibles dans un même échantillon.
Liens et Articles Scientifiques
-
Article de base sur la PCR: Mullis et Faloona, 1987
-
PCR en temps réel (qPCR): Heid et al., 1996
-
Applications médicales de la PCR: Kalayjian et al., 1993
-
Optimisation des conditions de PCR: Dieffenbach et Dveksler, 1995
-
Utilisation de la PCR en médecine légale: Gill et al., 1985