Séquençage de l'ADN
-
Définition
Le séquençage de l'ADN (acide désoxyribonucléique) consiste à déterminer la succession linéaire des bases A, C, G et T qui composent la structure de l'ADN. Il est intéressant d'avoir la séquence de l'ADN car on a accès à toutes les informations qui la contiennent (Lamoril, J. et al. 2008). On peut
-
Applications
-
Historique
-
Techniques
Les deux premières techniques sont celle de Maxam-Gilbert et celle de Sanger, Elles étaient décrites en 1977, Il existe aussi d’autres techniques modernes.
Quelques techniques et leurs méthode de fonctionnement :
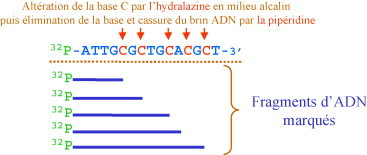
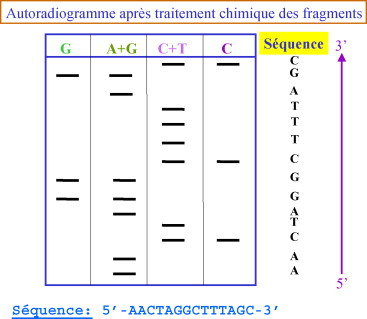
- Technique de Maxam-Gilbert: C’est une méthode chimique de séquençage,Les réactifs clivent spécifiquement après chacune des bases A, C, G, [A + G], [C + T]. Cette technique est basée sur la propriété de certains agents chimiques, l’hydrazine, le diméthyl sulfate (DMS) et l’acide formique, de modifier les bases de l’ADN. Dans un second temps, la pipéridine est ajoutée et « casse » les brins d’ADN au niveau des bases modifiées (Figure-1) . Les agents chimiques sont utilisés dans des conditions telles qu’ils n’agissent qu’avec un faible pourcentage des bases de l’ADN étudié. L’ADN à séquencer est marqué à une extrémité. Le plus souvent, il s’agit d’un marqueur radioactif. Le produit de séquence est déposé sur un gel d’acrylamide, puis la séquence lue après autoradiographie (Figure-2). L’ADN étudié peut être simple ou double brin. Cette technique permettait d’analyser des fragments allant jusqu’à 500 pb.
Figure (1)
Figure (2)
· Technique de Sanger
· Technique Shotgun
· Principe de séquençage de grands génomes
· Amplification génome complet / whole genome amplification (WGA)
· Séquençage par hybridation
· Méthode de PCR sur colonies (dite amplification clonale) ou PCR colonies (polony)
· Pyroséquençage
· Séquençage par spectrométrie de masse