Séquençage de l'ADN
-
C'est quoi un ADN :
L'ADN, ou acide désoxyribonucléique, est une molécule présente dans toutes les cellules vivantes. C'est une macromolécule biologique qui contient les instructions génétiques essentielles nécessaires au développement, au fonctionnement, à la croissance et à la reproduction de tous les organismes vivants, qu'ils soient simples ou complexes.
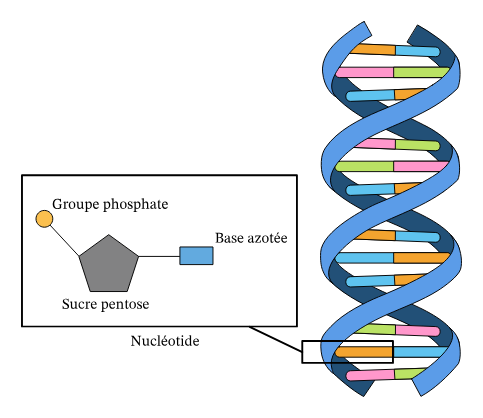
L'ADN se présente sous la forme d'une double hélice, semblable à une échelle enroulée, composée de deux brins complémentaires d'unités de base appelées nucléotides. Chaque nucléotide se compose d'un sucre (désoxyribose), d'un groupement phosphate et d'une base azotée. Les quatre bases azotées trouvées dans l'ADN sont l'adénine (A), la thymine (T), la cytosine (C) et la guanine (G). Ces bases se lient spécifiquement l'une à l'autre, avec l'adénine s'associant à la thymine et la cytosine s'associant à la guanine, formant ainsi les "barreaux" de l'échelle de la double hélice.
L'ADN contient les informations génétiques codées sous forme de séquences de nucléotides. Ces informations sont transmises d'une génération à l'autre et sont utilisées par les cellules pour fabriquer des protéines, qui sont les principaux acteurs de nombreuses fonctions biologiques dans les organismes vivants.
Cliquez sur la photo pour voir une vidéo explicative
-
Définition du séquençage d'ADN:
Le séquençage de l'ADN (acide désoxyribonucléique) consiste à déterminer la succession linéaire des bases A, C, G et T qui composent la structure de l'ADN. Il est intéressant d'avoir la séquence de l'ADN car on a accès à toutes les informations qui la contiennent (Lamoril, J. et al. 2008).
-
Applications
Le séquençage est une technique essentielle avec diverses applications. En médecine, il est utilisé pour diagnostiquer les maladies génétiques et les mutations associées aux cancers, ce qui guide les traitements. En recherche génétique, il aide à comprendre la structure et la fonction des gènes. La pharmacogénomique exploite les variations génétiques pour personnaliser les traitements. Les microbiomes et la génomique environnementale révèlent la diversité génétique des organismes et leur interaction avec leur environnement. Le séquençage de novo permet d'étudier des organismes sans séquence de référence. L'évolution est étudiée en comparant les séquences génétiques entre espèces ou populations. En agronomie, il est utilisé pour améliorer les plantes et les animaux d'élevage. Ces applications illustrent la polyvalence du séquençage dans divers domaines de recherche et de médecine.
-
Historique
Le séquençage de l'ADN est inventé dans la deuxième moitié des années 1970. Deux méthodes sont développées indépendamment, l'une par l'équipe de Walter Gilbert, aux États-Unis, et l'autre par celle de Frederick Sanger (en 1977), au Royaume-Uni, Pour cette découverte, Gilbert et Sanger sont récompensés par le prix Nobel de chimie en 1980.
-
Techniques
Les deux premières techniques sont celle de Maxam-Gilbert et celle de Sanger, Elles étaient décrites en 1977, Il existe aussi d’autres techniques modernes.
Voici quelques techniques avec leurs méthode de fonctionnement :
- Technique de Maxam-Gilbert: C’est une méthode chimique de séquençage,Les réactifs clivent spécifiquement après chacune des bases A, C, G, [A + G], [C + T]. Cette technique est basée sur la propriété de certains agents chimiques, l’hydrazine, le diméthyl sulfate (DMS) et l’acide formique, de modifier les bases de l’ADN. Dans un second temps, la pipéridine est ajoutée et « casse » les brins d’ADN au niveau des bases modifiées (Figure-1) . Les agents chimiques sont utilisés dans des conditions telles qu’ils n’agissent qu’avec un faible pourcentage des bases de l’ADN étudié. L’ADN à séquencer est marqué à une extrémité. Le plus souvent, il s’agit d’un marqueur radioactif. Le produit de séquence est déposé sur un gel d’acrylamide, puis la séquence lue après autoradiographie (Figure-2). L’ADN étudié peut être simple ou double brin. Cette technique permettait d’analyser des fragments allant jusqu’à 500 pb.
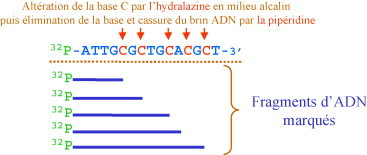
Figure (1)
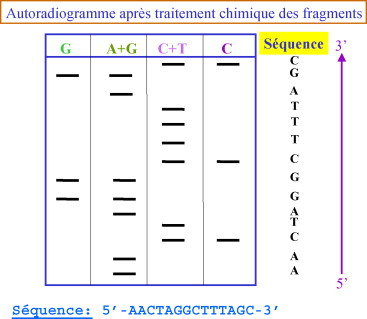
Figure (2)
· Technique de Sanger: Elle a en effet rapidement dépassé la méthode de Maxam-Gilbert pour la remplacer et reste à ce jour la principale méthode de séquençage utilisée dans les laboratoires. Cependant, elle reste limiter car elle ne peut sequencer que des fragments entre 400-800pb. Son principe est le suivant.
-
Préparation de l'ADN: Tout d'abord, l'ADN à séquencer est préparé. Il peut s'agir d'un fragment d'ADN spécifique ou d'une séquence plus longue.
-
PCR : L'ADN est amplifié par PCR pour obtenir suffisamment de matériel génétique pour l'analyse. Puis, il faudra dénaturer les échantillons obtenus pour obtenir un ADN simple brin.
- Réaction de séquençage: A partir de notre ADN, on obtient un ADNc marqué grâce au mélange réactionnel qui contient les tampons, l’ADN polymérase, des déoxynucléotides triphosphates (dNTP, dA-, dC-, dG-, dT-TP) mais aussi des didéoxynucléotides triphosphates (ddNTP, ddA-, ddC-,ddG-, ddT-TP) couplés à un fluorophore différents. Lors de cette réaction, il y a statistiquement au moins une chance pour chaque base qu'un ddNTP soit attribué à la place d'un dNTP, et arrêtera ainsi l'action de l'ADN polymérase. Nous obtiendrons alors des échantillons de taille différentes. (Figure 3)
- Électrophorèse: De nos jours, il faut utiliser un automate de séquençage pour effectuer une électrophorèse de capillaire (sinon le travail est fastidieux).
-
Analyse des données: Les données de séquençage sont ensuite analysées par ordinateur pour assembler la séquence complète et déterminer l'ordre précis des nucléotides dans l'ADN.
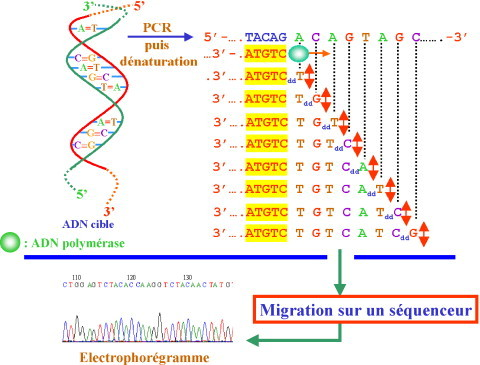
Figure (3)
· Séquençage par hybridation :
Le séquençage par hybridation repose sur l’utilisation de puces à ADN contenant de plusieurs centaines (pour les puces de première génération) à plusieurs milliers d’oligonucléotides (courtes chaînes de nucléotides) . L’ADN à analyser est coupé en de multiples fragments qui sont ensuite incubés sur la puce où ils vont s’hybrider avec les oligonucléotides dont ils sont complémentaires. La lecture de la puce (la détection des oligonucléotides hybridés), permet d’obtenir le spectre de la séquence d’ADN, c’est-à-dire sa composition en sous-séquences de n nucléotides, où n est la taille des sondes sur la puce utilisée. Le traitement informatique du spectre permet ensuite de reconstituer la séquence entière. (Figure-4)
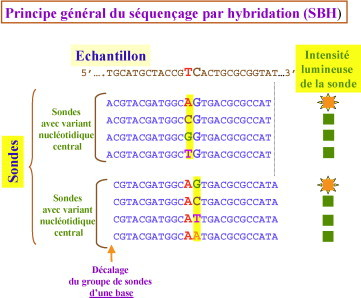
Figure (4)
· Technique Shotgun :
La technique de Shotgun sequencing consiste brièvement à fragmenter l'ADN en petits morceaux, à séquencer chaque fragment individuellement, puis à assembler les séquences pour reconstruire le génome ou le fragment d'ADN d'origine.
· Principe de séquençage de grands génomes:
L’automatisation à l’aide de robots a permis de séquencer de nombreux génomes dont le génome humain. Pour réaliser le séquençage d’un grand génome, une carte physique du génome est d’abord constituée.
· Amplification génome complet / whole genome amplification (WGA) :
La WGA est une méthode qui permet d'amplifier l'ensemble du génome à partir d'un échantillon biologique, fournissant ainsi suffisamment d'ADN pour les analyses ultérieures, même à partir d'échantillons de faible quantité ou de mauvaise qualité.
· Pyroséquençage :
Le pyroséquençage est une méthode basée sur la détection de la libération de pyrophosphate qui a lieu lors de l'incorporation de nucléotides. Le brin d'ADN doit d'abord être amplifié par PCR avant le séquençage. L'ordre dans lequel les nucléotides seront ajoutés est ensuite choisi dans le séquenceur (c'est-à-dire G-A-T-C). Chaque fois qu'un nucléotide spécifique est ajouté dans la chaîne par l'ADN polymérase, un pyrophosphate est libéré et converti en ATP par une ATP sulfurylase. L'ATP stimule alors l'oxydation de la luciférine en luciférase, cette réaction génère un signal lumineux enregistré sous la forme d'un pic. L'incorporation de chaque nucléotide est alors corrélée à un signal. Le signal lumineux est proportionnel à la quantité de nucléotides incorporés lors de la synthèse du brin d'ADN (deux nucléotides incorporés correspondent à deux pics). Lorsque les nucléotides ajoutés ne sont pas incorporés dans la molécule d'ADN, aucun signal n'est enregistré. Cette technique ne nécessite ni nucléotides marqués par fluorescence ni électrophorèse sur gel.
· Séquençage par spectrométrie de masse :
La spectrométrie de masse pour le séquençage fragmente l'ADN, ionise les fragments, analyse leur masse pour reconstruire la séquence. Bien qu'alternative, elle est limitée en taille de fragments et précision, souvent combinée à d'autres méthodes pour des résultats plus fiables.
- Séquençage par nanopore :
Le séquençage de l'ADN par les nanopores est une méthode utilisée depuis 1995. Elle permet de déterminer l'ordre dans lequel les nucléotides sont disposés sur un fragment d'ADN donné à l'aide de nanopores (trou avec un diamètre de l'ordre du nanomètre).
Des protéines cellulaires transmembranaires possédant des petits pores peuvent agir comme des nanopores. Des nanopores peuvent également être construits dans un résidu de silicium simplement en y effectuant un trou d'une dizaine de nanomètres. Le morceau de silicium est par la suite rempli lentement par sculptage à l'aide de faisceau d'ions.
La méthode nanopore est une technique de séquençage d'ADN basée sur l'utilisation de nanopores, de minuscules trous à l'échelle nanométrique. Méthodologie:
-
-
Préparation de l'échantillon: L'ADN à séquencer est préparé et dénaturé pour obtenir des brins simples.
-
Passage à travers le nanopore: Les brins d'ADN sont acheminés à travers un nanopore intégré dans une membrane, généralement en silicium ou en polymère. Un potentiel électrique est appliqué à travers la membrane, ce qui entraîne le passage des ions à travers le nanopore.
-
Mesure de la conductance ionique: Pendant que l'ADN traverse le nanopore, il perturbe le flux d'ions à travers celui-ci. Cette perturbation est mesurée sous forme de variations de conductance ionique, ce qui permet de déduire des informations sur la séquence d'ADN.
-
Analyse des signaux: Les variations de conductance ionique sont enregistrées et analysées pour reconstruire la séquence d'ADN. Chaque base nucléotidique produit une signature caractéristique de conductance ionique, permettant ainsi l'identification de la séquence.
-
La méthode nanopore présente plusieurs avantages, notamment sa rapidité, sa simplicité et sa capacité à séquencer de longues séquences d'ADN sans nécessiter d'amplification préalable. Elle est utilisée dans divers domaines de la recherche en génétique, en médecine et en biologie, ainsi que dans des applications de diagnostic et de surveillance environnementale.
-
Conclusion:
Le séquençage de l’ADN par la méthode de Sanger est actuellement la méthode de choix dans les laboratoires hospitaliers et de recherche . Sans cesse améliorée depuis plus d’une dizaine d’années, elle semble atteindre aujourd’hui ses limites même si des améliorations, notamment la miniaturisation de cette technique sont en développement . Outre les limites technologiques, le coût du séquençage selon cette méthode est encore élevé .
La méthode nanopore commercialisée à partir de 2016 est aussi souvent utilisée vu les avantages qu'elle offre.
-
Bibliographie :
https://www.ncbi.nlm.nih.gov/pmc/articles/PMC7147846/
https://fr.wikipedia.org/wiki/S%C3%A9quen%C3%A7age_de_l%27ADN#
https://fr.wikipedia.org/wiki/S%C3%A9quen%C3%A7age_par_nanopores#
No Comments